Transformación bacteriana: Un proceso biológico revolucionario

La transformación de organismos (o transfección en caso de células de mamíferos) para la producción de proteínas recombinantes es una herramienta revolucionaría que ha cambiado por completo la producción de medicamentos, alimentos, vacunas, etc. Sobretodo, destaca la transformación de bacterias por su plétora de usos en la ingeniería genética. En este proceso de transformación, se introduce ADN exógeno (perteneciente a un organismo diferente) en bacterias que codifica a proteínas específicas. De esta manera, se sintetizan diversos productos como la insulina para tratar la diabetes, lipasas para procesar lípidos en alimentos y eritropoyetina para tratar anemia, entre otros [1]. Para lograr la transformación de bacterias, se realizan los siguientes pasos:
Preparación del ADN recombinante
De manera natural, las bacterias pueden compartir entre ellas moléculas de ADN circular denominadas plásmidos en un proceso llamado conjugación [2]. Debido a la conjugación y que las bacterias no tienen un núcleo concreto dentro de sus células, el proceso de transformación es más sencillo que en organismos eucariotas, las cuales poseen un núcleo y organelos más complejos. Aprovechando estos fenómenos, solamente es necesario insertar un plásmido con el gen recombinante (material genético que proviene de un organismo distinto) para que la bacteria comience a sintetizar la proteína deseada.
Sin embargo, es imperativo establecer correctamente todos los elementos del plásmido para lograr una correcta expresión de la proteína, tal como se observa en la Figura 1:
Entre estos elementos, encontramos un origen de replicación para la duplicación del plásmido, un promotor o región de ADN que controla el inicio de la transcripción del gen, la secuencia del gen que codifica a la proteína insertada entre los sitios de restricción, un terminador que controla el final de la transcripción, un sitio de unión al ribosoma dónde el transcrito de ADN se traduce a proteína, así como otros elementos genéticos.
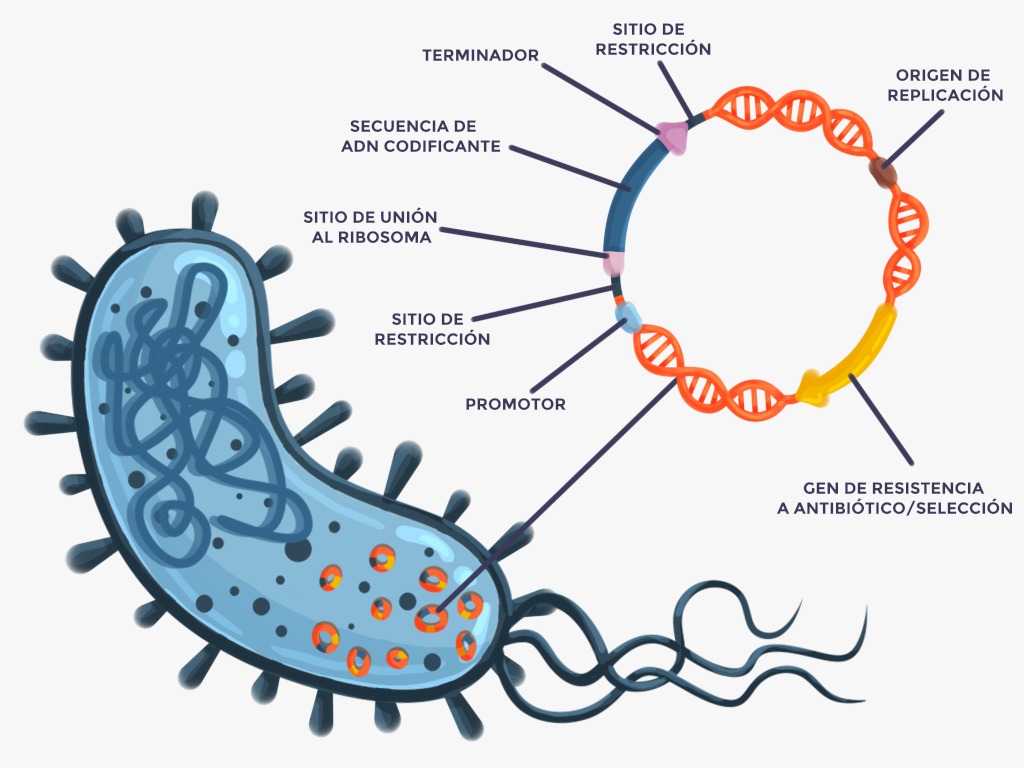
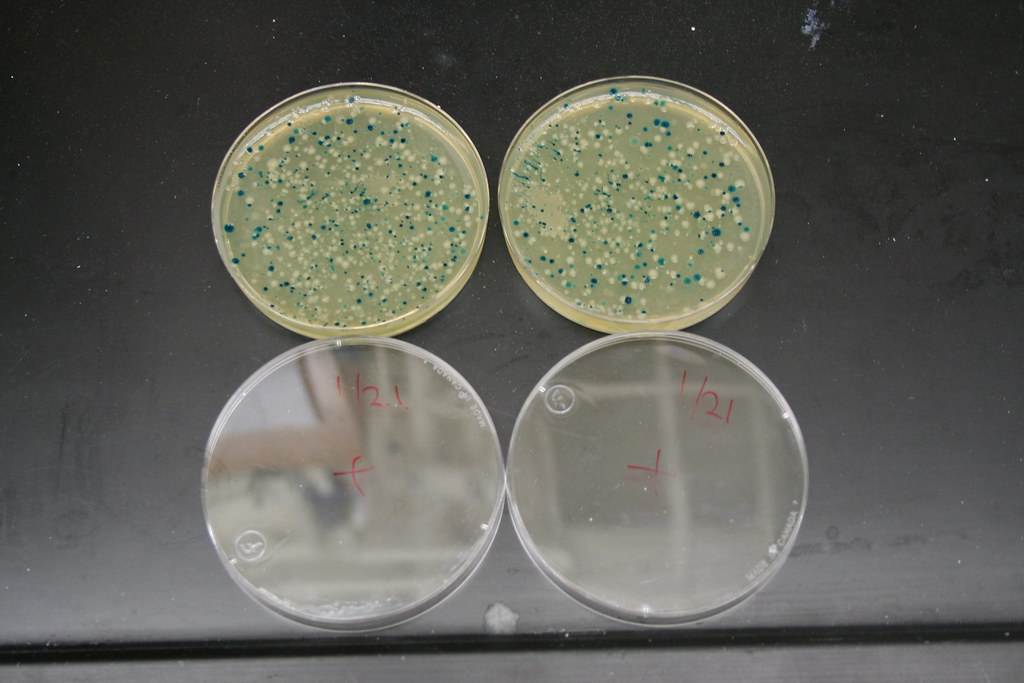
Asimismo, es importante que el plásmido contenga algún gen de selección para diferenciar a las bacterias transformadas de las no transformadas [3]. Este gen de selección puede ser un gen de resistencia a antibióticos como se muestra en la Figura 2, o un marcador como el gen de GFP que le proporciona luminiscencia a las bacterias transformadas [4]. También es importante utilizar células competentes, capaces de ser transformadas [5].
Transformación de bacterias por electroporación
La electroporación es un método en el que se emplean pulsos eléctricos de alto voltaje para crear poros en la membrana celular de las bacterias. A través de estos poros, puede entrar el ADN plasmídico. Las muestras con las bacterias mezcladas con el ADN plasmídico son colocadas en cubetas de electroporación con electrodos de cada lado. Las muestras entonces son colocadas en un electroporador. Dentro del aparato, las bacterias se someten a un campo eléctrico de hasta 10,000 V/cm, cambiando el voltaje de la membrana lipídica lo que conlleva a la formación de poros [5].
Transformación de bacterias por choque térmico
Normalmente, tanto la membrana celular y las moléculas de ADN están cargadas negativamente, por lo que a las muestras se les agregan moléculas de calcio. Los iones del calcio neutralizan las cargas de las moléculas para disipar las fuerzas de repulsión. Este procedimiento se realiza a la par de un choque térmico, en el que un súbito cambio de temperatura crea una diferencia de presión entre el interior y exterior de la célula, fomentando la formación de poros para que pueda internalizarse el ADN [6]. En ambos métodos, al regresar a condiciones normales, la membrana celular se regenera naturalmente.
La gran versatilidad de las bacterias para la producción de proteínas es sobresaliente. Sin embargo, no es la única forma de transformación genética que existe, además de tener ciertos incovenientes. Por ejemplo, las bacterias son incapaces de llevar a cabo modificaciones post-transcripcionales; es decir, solamente sintetizan la proteína tal cual como la codifica el ADN y no pueden cambiar la secuencia después. Por ello, al producir proteínas que requieren modificaciones post-traduccionales como la fosforilación o la acetilación, se realiza la transformación de organismos eucariotes, como las plantas, las levaduras o en algunos casos se realiza la transfección de células de mamíferos [7].
Aún así, la transformación bacteriana es una gran herramienta en el día a día del biotecnólogo. El mejor ejemplo de esto es la ya mencionada insulina. En los años previos, la producción de insulina recombinante en 1978 se obtenía del páncreas de diversos animales. Dicha insulina no lograba satisfacer la demanda y había mucha varianza entre la potencia de cada lote [8]. Hoy en día la insulina semisintética elaborada por métodos biotecnológicos como la transformación de E. coli, supera a la insulina animal purificada tanto en la demanda, además de tener una mejor absorción, menos degradación y menos reacciones en la piel al inyectar la insulina al cuerpo [9].
Referencias
- Khan, S., Ullah, M., Siddique, R., Nabi, G., Manan, S., Yousaf, M., & Hou, H. (2016). Role of Recombinant DNA Technology to Improve Life. International Journal of Genomics, 2016, 1–14. doi:10.1155/2016/2405954
- Shintani, M., Sanchez, Z. K., & Kimbara, K. (2015). Genomics of microbial plasmids: classification and identification based on replication and transfer systems and host taxonomy. Frontiers in Microbiology, 6. doi:10.3389/fmicb.2015.00242
- Alejo Cantoia, Dianela Aguilar Lucero, Eduardo A. Ceccarelli, & Germán L. Rosano. (2021). Chapter Two – From the notebook to recombinant protein production in Escherichia coli: Design of expression vectors and gene cloning. Methods in enzymology, 659, 19-35. https://doi.org/10.1016/bs.mie.2021.07.008
- Millán, I., & Becu-Villalobos, D. (2009). La proteína verde fluorescente ilumina la biociencia. Medicina (Buenos Aires), 69 (3). http://www.scielo.org.ar/scielo.php?script=sci_arttext&pid=S0025-76802009000400015&lng=es&tlng=es
- JoVE Science Education Database. (2023). Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: Electroporation. JoVE, Cambridge, MA. https://www.jove.com/es/v/5060/bacterial-transformation-electroporation
- JoVE Science Education Database. (2023). Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: The Heat Shock Method. JoVE, Cambridge, MA. https://app.jove.com/es/v/5059/bacterial-transformation-the-heat-shock-method
- Deribe, Y. L., Pawson, T., & Dikic, I. (2010). Post-translational modifications in signal integration. Nature Structural & Molecular Biology, 17(6), 666–672. doi:10.1038/nsmb.1842
- Mbanya, J. C., Sandow, J., Landgraf, W., & Owens, D. R. (2017). Recombinant Human Insulin in Global Diabetes Management – Focus on Clinical Efficacy. European Endocrinology, 13(01), 21. doi:10.17925/ee.2017.13.01.21
- Raptis S., & Dimitriadis G. Human insulin. (1985). Clin Physiol Biochem., 3 (1), 29-42. PMID: 3888492.
Fotografía: Cortesía de Fernan Federici Vía Flickr
Artículos más recientes

El resurgimiento de los Dinosaurios
27/Sep/2024

Expedición a los parques: bitácora de un explorador
25/Sep/2024

Ingeniería genética en los alimentos
23/Sep/2024
Día Mundial del Alzheimer: Una enfermedad degenerativa
21/Sep/2024

MÁQUINAS QUE PIENSAN
20/Sep/2024

¿Cual es la genética detrás de la gente Pelirroja?
18/Sep/2024

Desafiando la Fiebre Q: Diagnóstico con un nuevo Biosensor
10/Jul/2024